AI精准设计大环肽:打破药物靶点“不可成药”魔咒
- 2025-12-24
- 148

2025年6月20日,由诺贝尔奖获得者、蛋白质设计先驱David Baker教授领导的研究团队在《Nature Chemical Biology》杂志上发表了RFpeptides新成果,利用RFpeptides全新框架能够从头设计具有高亲和力靶向蛋白的大环肽,这是药物研发的一个新纪元。我们可能正站在一个新时代的起点—从依赖随机筛选和天然产物发现的传统模式,迈向精准计算引导的理性设计时代。
一、传统肽类药物研发的局限性
1.源头依赖与随机性
严重依赖对天然活性肽的修饰,创新不足;或从海量随机肽库中筛选,难度大。
2.高通量筛选的资源密集型挑战
依赖噬菌体展示等技术,需对数万亿分子进行多轮实验筛选,才能分离出极少数的有活性的苗头分子。
3.大环肽设计能力薄弱
难以理性控制肽的环化与三维构象,环化后易失去活性,系统性设计高亲和力大环肽极具挑战。
4.难以攻克“不可成药”靶点
面对缺乏深结合口袋的蛋白质靶点,传统随机筛选方法成功率极低,无法实现有目的的理性设计。
二、大环肽与AI设计成为破局“不可成药”的新纪元
生物制剂虽然能够以高亲和力和高选择性结合多种治疗靶点,但由于其分子量大、极性高,通常无法穿过细胞膜,这限制了它们只能作用于细胞外靶点。相反,小分子能够进入细胞内靶点,但不适合靶向缺乏深疏水口袋的蛋白质。原则上,可以开发出大小介于小分子和蛋白质之间的大环肽,以调节传统治疗方式无法触及的分子靶点。然而,其理性设计一直是无解难题。
David Baker团队开发的RFpeptides框架,结合RoseTTAFold2精准预测和生成式AI的创造能力,实现了从“筛选”到“设计”的根本转变。它不再依赖随机库,而是直接根据靶蛋白的3D结构,逆向生成与之相匹配的大环肽序列,从而开启将药物设计从不确定性推向可预测、可编程的数字化设计新纪元。
三、计算模型的精确复现
RFpeptides是一种生成式深度学习(DL)流程,可用于针对多种蛋白质靶标精确从头设计大环结合剂。与传统的设计方法相比,RFpeptides具有多个优势:
1.该设计方法能够更快、更高效地发现大环结合剂。尽管每个靶标测试的设计候选物不到20个(相比之下,传统的基于文库的方法测试了数万亿个肽),但我们无需任何进一步的实验优化就为两个靶标获得了高亲和力结合剂,成功率远高于以往任何方法。
2.与基于随机文库的传统方法的非靶向性不同,RFpeptides可用于设计针对特定斑块和位点的定制结合剂。
3.设计模型的原子级精确性使得能够进行结构引导的优化,以进一步提高亲和力,从而绕过复杂结构确定的瓶颈。结合膜穿透的设计原则,RFpeptides能够设计出同时优化靶标结合和细胞通透性或口服生物利用度的肽。
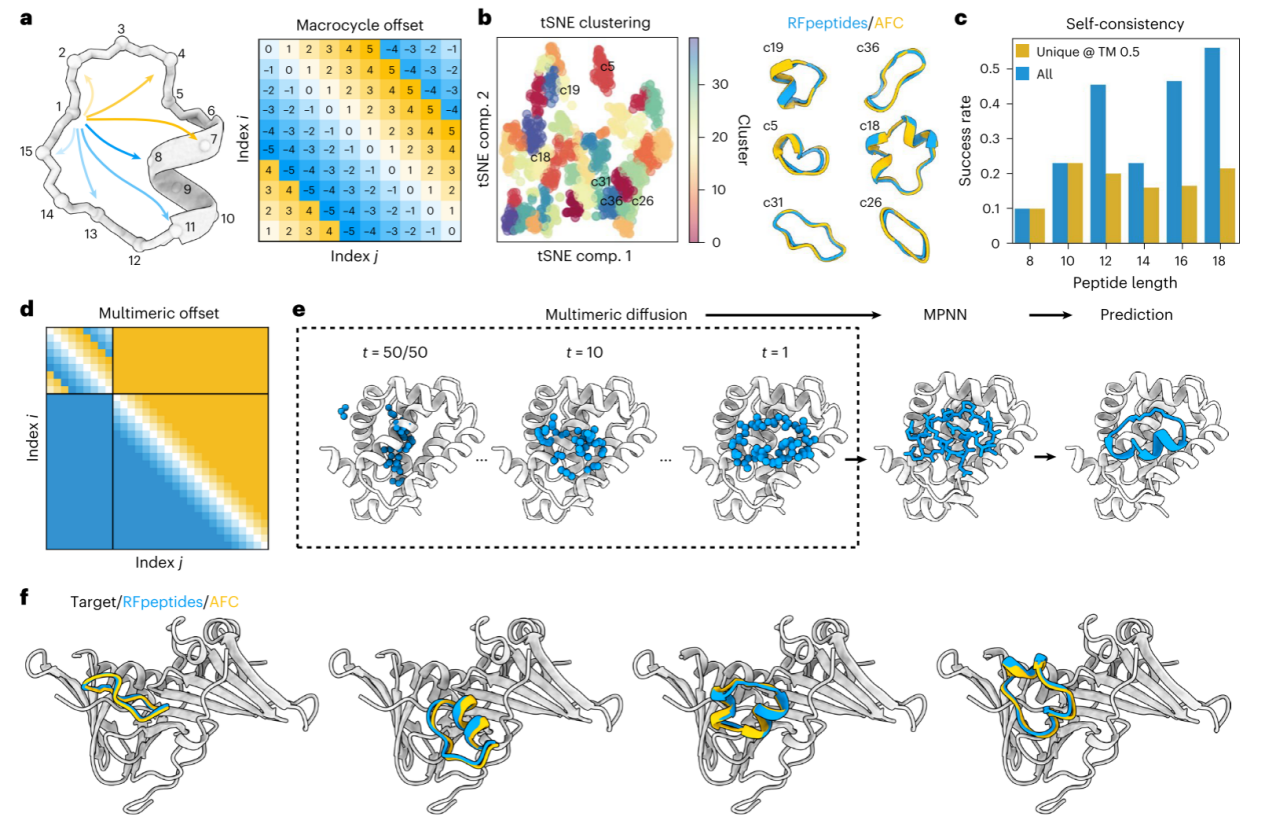
图1 RFpeptides技术框架图
图1首先构建了RFpeptides的技术框架,揭示其如何通过循环相对位置编码改造RoseTTAFold2(RF2)和RFdiffusion模型,实现大环肽主链的高效生成。
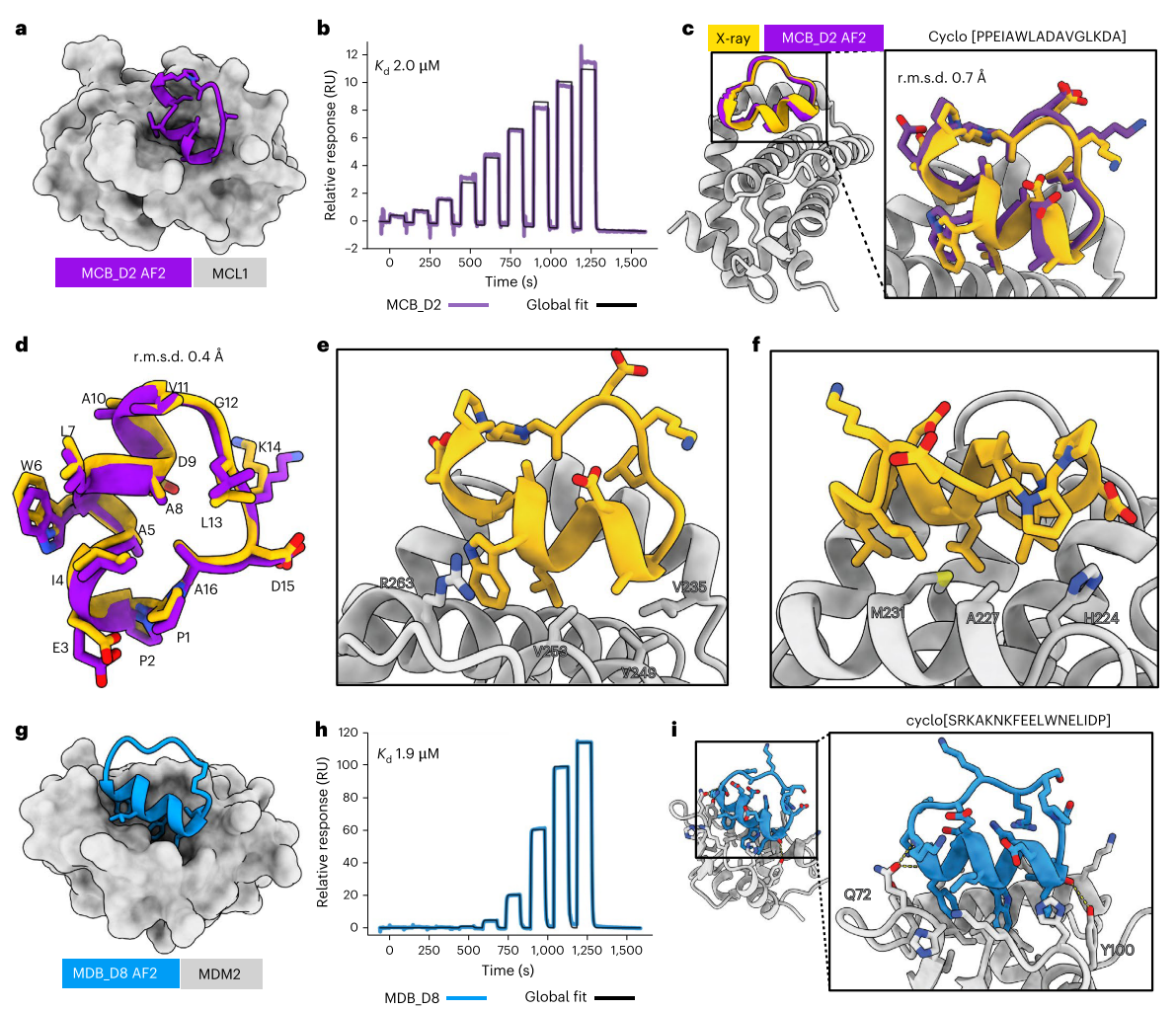
在此基础上,图2针对MCL1和MDM2两个经典抗癌靶点展开验证,二者均为调控细胞存活的关键蛋白,且结合口袋以α-螺旋为主。针对MCL1设计的MCB_D2,经X射线晶体结构证实,其与靶标的Cα RMSD仅0.7Å(主链单独比对0.4Å),侧链构象与设计模型高度一致,SPR检测显示结合亲和力达2.0μM,核心结合作用不仅包括螺旋区疏水相互作用,还涉及环区独特的阳离子-π作用;针对MDM2的MDB_D8则以1.9μM的亲和力结合靶标,其关键作用基序与天然 MDM2-p53复合物相似,进一步证明RFpeptides 在明确结构靶点上的精准设计能力。
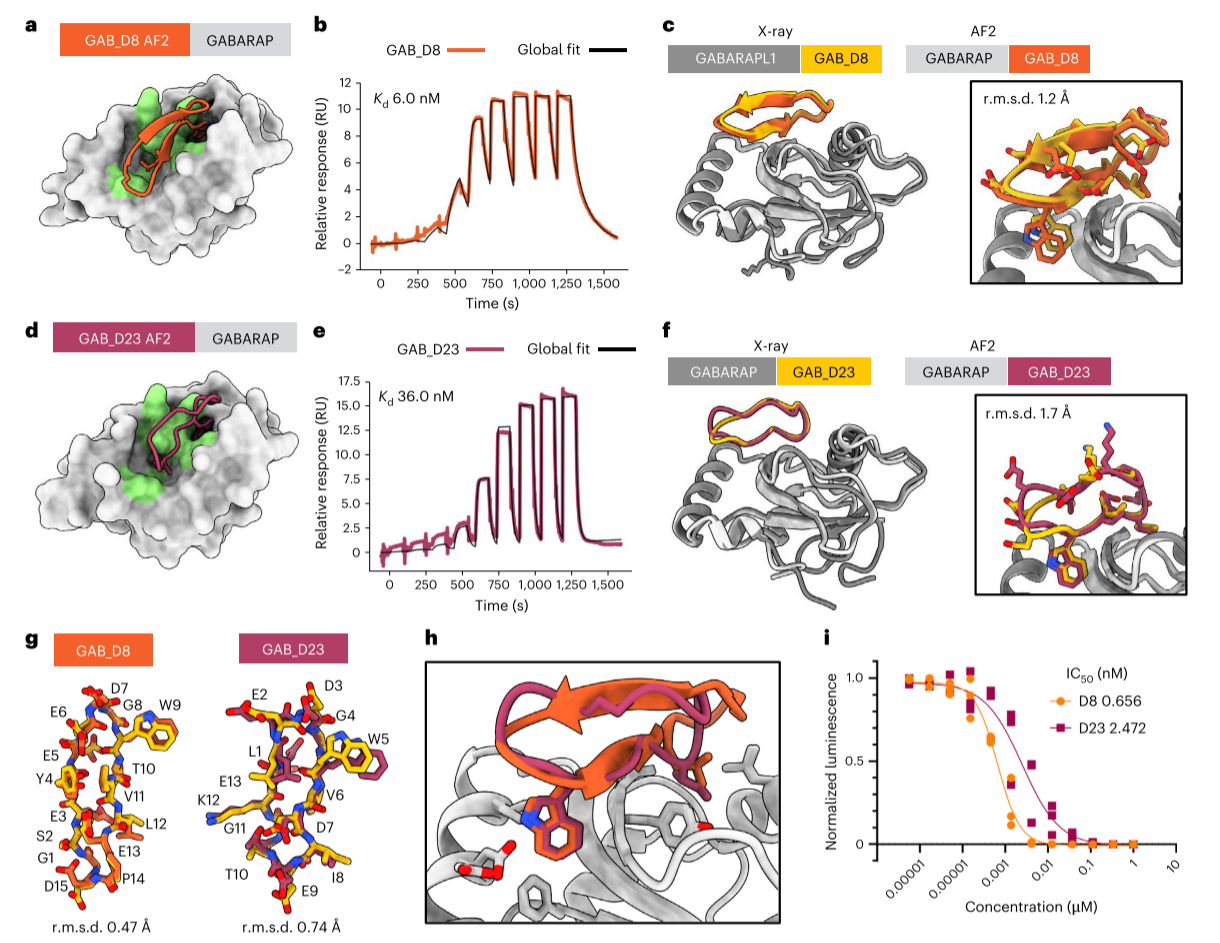
图3 针对髓系细胞白血病1和MDM2的大环结合剂的从头设计图
当靶点结合口袋结构更为复杂时,RFpeptides的优势进一步凸显,这一点在图3中得到充分体现。GABARAP作为自噬调控关键蛋白,是癌症与神经疾病治疗的潜在靶点,其复杂口袋结构对设计方法提出更高要求。SPR检测结果显示,设计的GAB_D8和GAB_D23的Kd值分别低至6nM和36nM,AlphaScreen实验中IC50值仅0.7nM和2.5nM,其中GAB_D8更是目前已知最有效的GABARAP大环结合剂。X射线晶体结构验证显示,GAB_D8与同源蛋白GABARAPL1结合的Cα RMSD为1.2Å(单独比对0.47Å),GAB_D23与GABARAP结合的对应RMSD为1.7Å(单独比对0.74Å),并且多序列比对(MSA)结果表明优化的预测模型与实验结构匹配度更高,证实该方法能应对非单一结构类型的复杂靶点。
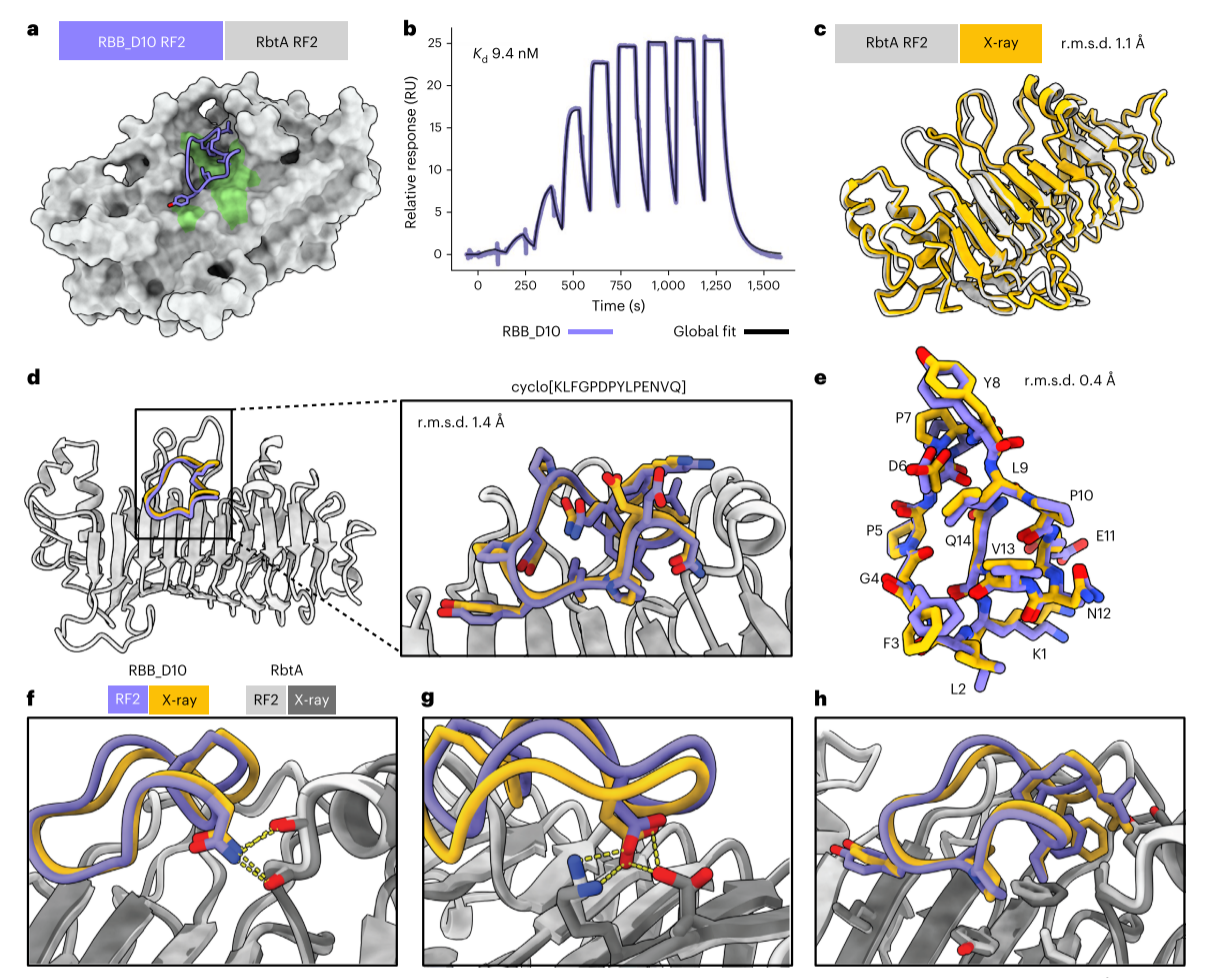
图4 针对鲍曼不动杆菌RbtA的环肽结合剂的从头设计图
图4突破了“依赖已知实验结构”的传统设计局限,针对无任何实验结构的鲍曼不动杆菌表面蛋白RbtA展开设计。研究先通过AF2 和RF2预测其结构,再基于预测结构设计大环结合剂RBB_D10。
实验结果显示,RBB_D10不仅能靶向传统方法难以应对的平坦蛋白表面,还展现出9.4nM的高结合亲和力;X射线晶体结构进一步证实,RbtA实验结构与预测模型的Cα RMSD为1.1-1.2Å,RBB_D10与靶标结合的结构与设计模型比对,Cα RMSD仅1.4Å(主链单独比对 0.4Å),侧链相互作用完全符合设计预期。这一成果彻底打破了靶点结构未知的技术瓶颈,为新型致病菌靶点或“不可成药”靶点的药物研发提供了全新路径。
四、结语
综上,RFpeptides 通过“模型改造-明确结构靶点验证-复杂结构靶点突破-未知结构靶点拓展”的层层递进验证,构建了一套独特的大环化合物设计体系,可应用于不同结构类型的靶点。其核心优势在于将DL结构生成能力与蛋白质-配体相互作用的精准预测相结合,不仅解决了传统筛选方法效率低的问题,又突破了现有计算设计方法对已知实验数据的依赖,加速推进大环肽类诊断试剂与治疗药物的开发进程。
卡梅德生物(KMD Bioscience)依托成熟的重组蛋白表达及抗体发现等核心平台,可高效匹配AI设计所需的靶点蛋白制备与活性验证需求,结合噬菌体文库构建等传统技术,形成“AI设计-实验验证-工艺优化”的完整闭环,为攻克“不可成药”靶点提供从设计到落地的一站式服务,助力RFpeptides 技术快速转化为实际研发成果。
引用文献:
[1] Rettie SA, Juergens D, Adebomi V, et al. Accurate de novo design of high-affinity protein-binding macrocycles using deep learning. Nat Chem Biol. Published online June 20, 2025.
[2] Bhardwaj G, O'Connor J, Rettie S, et al. Accurate de novo design of membrane-traversing macrocycles. Cell. 2022;185(19):3520-3532.e26.
[3] Zhang H, Ginn J, Zhan W, et al. Design, Synthesis, and Optimization of Macrocyclic Peptides as Species-Selective Antimalaria Proteasome Inhibitors. J Med Chem. 2022;65(13):9350-9375.
- 大环肽
- AI设计
- 多肽筛选
- 药物研发
-
 400-621-6806
400-621-6806 -
 13116038708(微信同号)
13116038708(微信同号) -
 周一至周五 8:30-18:00
周一至周五 8:30-18:00 -

-
 0
0
-





